Em 1981, foi relatado o primeiro gene clonado que codifica um cristal proteico de Bacillus thuringiensis (SCHNEPF; WHITELEY, 1981). Nos anos seguintes mais genes foram clonados, portanto, uma nomenclatura para as proteínas codificadas foi proposta (HOFTE; WHITELEY, 1989). Nesta nomenclatura, as proteínas Bt foram classificadas de acordo com os organismos-alvo: Cry I, tóxica para insetos da ordem Lepidoptera; Cry II, tóxica para Lepidoptera e Diptera; Cry III, com ação sobre insetos da ordem Coleoptera; e Cry IV, efetiva contra a ordem Diptera.
Apesar dessa nomenclatura ter se mostrado extremamente útil, logo demostrou limitações. Certas proteínas que compartilhavam sequências semelhantes muitas vezes tinham diferentes especificações, exigindo serem colocadas em diferentes grupos de classificação primária. Outro empecilho era a necessidade de obter-se dados abrangentes de bioensaios antes de uma proteína ser classificada.
Para superar esses desafios, em 1998 foi introduzida uma revisão na nomenclatura, classificando as proteínas apenas por similaridade de aminoácidos (CRICKMORE et al., 1998). Nesse sistema, foi adotada uma nomenclatura de quatro níveis, na qual as proteínas que compartilhavam pelo menos 45% de identidade de sequência foram colocadas no mesmo grupo de classificação primária (Cry1, Cry2 etc). Esta nomenclatura seguiu sendo usada por mais de 20 anos.
Figura 1. Classificação das proteínas Bt de acordo com a similaridade da sequência de amionoácidos.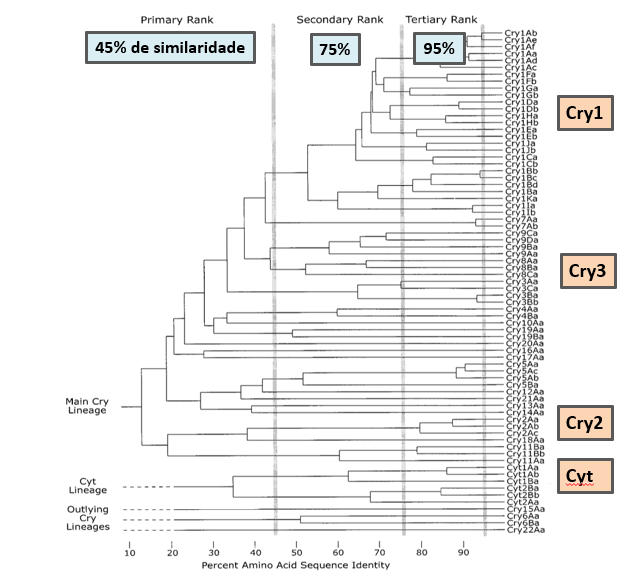
Fonte: Crickmore et al., 1998.
Ainda no ano de 1998, foi reconhecido que haviam diferentes tipos de proteínas Bt, sendo definidas duas denominações: Cyt para proteínas com atividade citolítica in vitro e ação sobre dípteros, e Cry para as demais proteínas inseticidas com conformação cristalina. Além disso, uma terceira sigla (Vip) também foi introduzida, para identificar proteínas inseticidas secretadas durante o crescimento vegetativo de B. thuringiensis e desprovidas da capacidade de esporulação (ESTRUCH et al., 1996).
Figura 2. Ordens de insetos controladas pelas proteínas Cry e Cyt.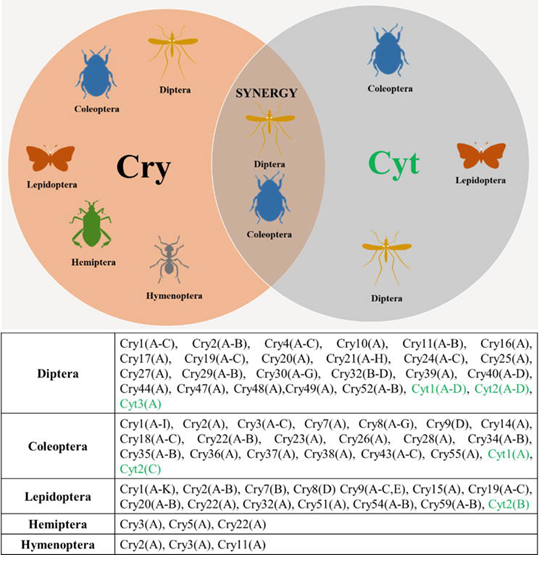
Fonte: Fernández-Chapa et al., 2019
Entretanto, com os avanços na área da biologia molecular, percebeu-se que há uma grande variedade de proteínas inseticidas derivadas de bactérias e que a nomenclatura existente restringia a apreciação de sua diversidade. Por esta razão, iniciou-se o desenvolvimento de novo um sistema de classificação, baseado tanto nas sequências de aminoácidos quanto nas suas configurações estruturais (CRICKMORE et al., 2020).
Inicialmente, foram definidas 16 classes estruturais. Três delas (Cry, Cyt e Vip3) não sofreram alterações em relação à nomenclatura anterior, enquanto novas proteínas (já descobertas ou que venham a ser identificadas no futuro) são nomeadas de acordo com a sequência de correspondência mais próxima, utilizando o grau de identidade para derivar o novo nome. Os níveis de segregação utilizados permaneceram muito semelhantes aos descritos em 1998: 45%, 75% e 95% de similaridade entre sequências para o primeiro, segundo e terceiro nível de classificação, respectivamente.
Figura 3. Estruturas representativas das diferentes classes de proteínas Bt.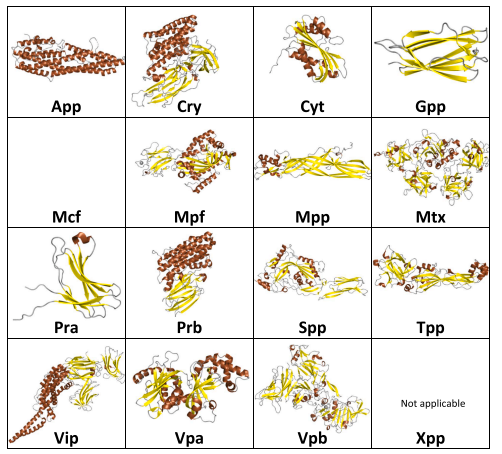
Fonte: CRICKMORE et al., 2020.
Segundo Crickmore et al. (2020), o novo sistema de classificação manteve o conceito original de que o papel mais importante da nomenclatura é fornecer um identificador único para cada proteína, facilitando tanto a pesquisa científica quanto a aplicação prática das tecnologias Bt. Com o avanço das técnicas de transgenia em diferentes culturas agrícolas e expansão da área de cultivo de variedades Bt, é provável que novas proteínas inseticidas sejam introduzidas no mercado, denotando a importância de classificá-las corretamente.
Para saber mais sobre o papel das diferentes proteínas Bt no controle de pragas do milho, clique aqui;
Revisão: Henrique Pozebon, doutorando PPGAgro – UFSM e Prof. Jonas Arnemann, PhD. e Coordenador do Grupo de Manejo e Genética de Pragas – UFSM
REFERÊNCIAS:
CRICKMORE, Neil et al. Revision of the nomenclature for the Bacillus thuringiensis pesticidal crystal proteins. Microbiology and molecular biology reviews, v. 62, n. 3, p. 807-813, 1998.
CRICKMORE, Neil et al. A structure-based nomenclature for Bacillus thuringiensis and other bacteria-derived pesticidal proteins. Journal of invertebrate pathology, p. 107438, 2020.
FERNÁNDEZ-CHAPA, D.; RAMÍREZ-VILLALOBOS, J.; GALÁN-WONG, L. Toxic Potential of Bacillus thuringiensis: An Overview, Protecting Rice Grains in the Post-Genomic Era, Yulin Jia, IntechOpen, 2019. Disponível em: https://www.intechopen.com/chapters/67369
ESTRUCH, Juan J. et al. Vip3A, a novel Bacillus thuringiensis vegetative insecticidal protein with a wide spectrum of activities against lepidopteran insects. Proceedings of the National Academy of Sciences, v. 93, n. 11, p. 5389-5394, 1996.
SCHNEPF, H. Ernest; WHITELEY, H. R. Cloning and expression of the Bacillus thuringiensis crystal protein gene in Escherichia coli. Proceedings of the National Academy of Sciences, v. 78, n. 5, p. 2893-2897, 1981.





